J'ai des données avec un meilleur tirage de ligne. J'ai besoin de dessiner deux autres lignes. L'un doit avoir le double de la pente et l'autre doit avoir la moitié de la pente. Plus tard, je vais utiliser la région à façon différentielle des points de couleur à l'extérieur comme par: Conditionally colour data points outside of confidence bands in RDans R tracer deux lignes, avec des pentes doubles et la moitié de la valeur de la ligne de meilleur ajustement
ensemble de données Exemple:
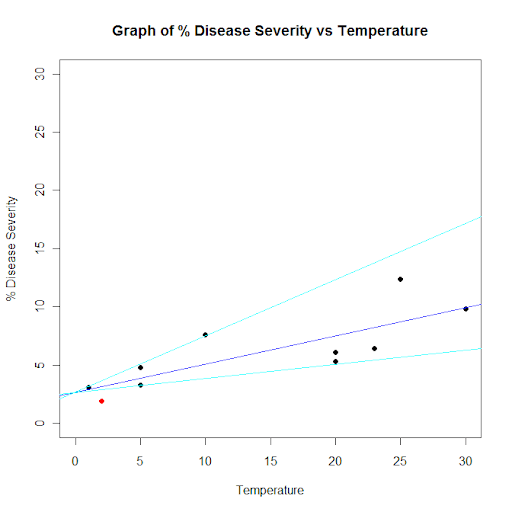
## Dataset from http://www.apsnet.org/education/advancedplantpath/topics/RModules/doc1/04_Linear_regression.html
## Disease severity as a function of temperature
# Response variable, disease severity
diseasesev<-c(1.9,3.1,3.3,4.8,5.3,6.1,6.4,7.6,9.8,12.4)
# Predictor variable, (Centigrade)
temperature<-c(2,1,5,5,20,20,23,10,30,25)
## For convenience, the data may be formatted into a dataframe
severity <- as.data.frame(cbind(diseasesev,temperature))
## Fit a linear model for the data and summarize the output from function lm()
severity.lm <- lm(diseasesev~temperature,data=severity)
# Take a look at the data
plot(
diseasesev~temperature,
data=severity,
xlab="Temperature",
ylab="% Disease Severity",
pch=16,
pty="s",
xlim=c(0,30),
ylim=c(0,30)
)
title(main="Graph of % Disease Severity vs Temperature")
par(new=TRUE) # don't start a new plot
abline(severity.lm, col="blue")
Est-ce vous êtes sûr que c'est ce que vous voulez vraiment faire? Vous réalisez que cette limite est fondamentalement insignifiante, n'est-ce pas? – hadley